长期以来,计算是科学研究中理论和实验之外非常重要的一个组成部分,比如在生命科学领域,基因测序数据分析、蛋白质结构解析和预测、药物筛选和药物设计等研究中,计算都扮演了非常重要的角色。随着AI技术的兴起,如何把AI技术应用到科学研究之中,来加速研究创新,是当前业界一个非常火热的话题。
AI+Science领域在近两年取得了非常多的进展和成果,其中最典型的例子就是基于人工智能方法的蛋白质结构预测算法AlphaFold2,它在CASP14蛋白质结构预测竞赛中实现了远超之前算法的精度,对绝大多数蛋白质结构的预测都能和实验结果足够接近,极大地促进了蛋白质结构研究领域的繁荣。除此之外,AI算法在分子动力学模拟、基因序列比对等领域也有较多的应用。
AI+Science的研究在大多数场景下都融合了AI计算和科学计算两种不同的计算需求,其中AI计算大多依赖于GPU,对计算精度要求低,而科学计算的数值精度要求更高,大多使用大量的CPU节点进行MPI通信来并行计算,当然现在也出现了大量借助GPU来进行加速计算的应用。因此,一款能够同时满足AI和科学计算两种应用需求的服务器产品,对于AI+Science这一应用场景,是十分必要的。
最近笔者拿到了一台当前业界顶级的GPU服务器浪潮NF5468A5,搭载了AMD 7543 CPU和NVIDIA A100 GPU,在CPU和GPU的技术指标都能算得上是业界顶配,完美地契合了AI+Science这一场景的需求。本文主要基于这一产品,对AlphaFold2,以及分子动力学领域的典型应用NAMD进行了性能测试,供相关领域人士参考。
一、测试平台介绍
NF5468A5是一款4U高度支持8块双宽GPU卡、搭配2颗AMD EPYC处理器,面向AI训练、AI推理、HPC、视频处理等多种应用场景的GPU服务器。
本次拿到的样机采用如下配置:
|
型号 |
NF5468A5 |
|
CPU |
2*AMD 7543,32核2.8 GHz主频,L3 Cache 256MB |
|
内存 |
512GB,16*32GB DDR4 3200ER |
|
硬盘 |
4*3.84TB NVME U.2 |
|
加速卡 |
8*NVIDIA A100 40GB GPU |
|
电源 |
4*2200W |

浪潮NF5468A5服务器
NF5468A5采用分区散热设计,内置独立的CPU和GPU主板,且分别安装在不同的平面。从机箱内部结构来看,设计者将发热量高的的GPU放在了机箱上3U空间,CPU板处于机箱下1U,内置6对12个6056风扇模组配合导风罩,实现机箱内风道分流,正是基于这种独特分层散热设计,使得送测的NF5468A5服务器可以支持2颗280W CPU+8颗300W的GPU。系统设计支持32条DDR4 ECC内存,支持LRDIMM/RDIMM,提供高达8T的本地内存,实现与超大L3 cache间的高速数据交互。

浪潮NF5468A5开箱俯视图
从浪潮官网发布的信息来看,NF5468A5采用了CPU直连GPU架构设计,没用PCIe Switch转发,能保证更高的传输带宽和更低的通信延迟,另外CPU和GPU之前的通信是基于PCIe 4.0协议的,理论单向带宽可以达到32GB/s。对于需要大量从CPU往GPU搬运数据的场景,这个设计会相当有用。总的来说,这台服务器的规格还是相当高的。
二、性能测评
在本次测试中,我们选取了AI+Science典型应用AlphaFold2和NAMD进行测试。在软件安装和运行中使用的操作系统、CUDA驱动如下表所示。
测试安装软件和对应版本
|
软件环境 |
软件版本 |
|
操作系统 |
Ubuntu20.04 |
|
CUDA驱动 |
CUDA 11.2 |
|
应用软件 |
AlphaFold2,NAMD V3.0 |
1. AlphaFold2测试
由DeepMind设计的AlphaFold2是一个端到端的蛋白质三维结构预测模型,可以从蛋白质序列直接预测蛋白质结构。其在CASP14竞赛上表现出了令人注目的准确性——GDT_TS为92.4,这意味着预测的大部分单体蛋白质结构准确度与实验足够接近,远超此前所有方法。在AlphaFold2的代码和模型开源之后,基于AlphaFold2的蛋白结构预测服务已经在高校、研究所和生物医药相关企业中广泛部署。
AlphaFold2的计算过程可分为数据处理和模型预测两部分,其中数据处理部分需要进行多序列比对(MSA)和模板搜索(template)过程,这部分的计算主要依赖于CPU的算力,因为这两个过程中需要比对和搜索的蛋白质序列和结构数据的总和达到了TB量级,因此整个计算过程特别耗时,且对磁盘IO也有较高的要求。同时整个比对和搜索过程的时长和蛋白序列长度正相关,对于长度达到数千个氨基酸的蛋白质序列,其搜索和比对过程可能长达数小时。
AlphaFold2的计算不仅需要GPU的算力,也对CPU算力以及磁盘IO性能等提出了很高要求。在本次测评中,我们基于浪潮NF5468A5服务器,同时配备4颗3.84T的浪潮NS8510G1 NVME磁盘来保证良好的IO性能,并配备了8张NVIDIA A100 40GB GPU。测试使用的AlphaFold2代码及模型来自DeepMind的github仓库:https://github.com/deepmind/alphafold。
本次测试我们选取了T1059(序列长度32)、T1037(序列长度404)和T1050(序列长度779)这3个不同长度的蛋白质序列进行测试。
下图是我们在测试过程中的数据处理部分的计算过程截图。
AlphaFold2数据处理部分计算过程截图
AlphaFold的模型预测阶段和其他深度学习模型的推理计算类似,是基于上一阶段得到的多序列比对结果和模板搜索结果作为模型输出推理得到蛋白质三维结构空间坐标的过程。这一过程主要在GPU上进行,因此GPU的算力对这一过程的快慢取到决定性的作用。在本次测试中我们选用了PCI-E 4.0接口的NVIDIA A100 40GB GPU,其算力属于当前业界一流水平。
AlphaFold2提供了5个不同的模型进行结构预测,这5个模型均是在原模型基础上采用不同的超参数finetuning得到的。最终的预测结果是在这5个模型的输出基础上基于打分模型遴选出一个最佳的模型结构。
下表为不同长度蛋白质序列在单张A100上预测得到的top1模型在不同阶段的计算性能结果。
|
Residuals |
running time for different stage (s) |
||||
|
features |
Process_features |
Predict&compile |
relax |
total |
|
|
32(T1059) |
847 |
1.0 |
50.9 |
7.6 |
906.5(15.1 mins) |
|
404(T1037) |
894 |
1.45 |
78.4 |
50.4 |
1024.25(17.1 mins) |
|
779(T1050) |
1160 |
6.23 |
207.9 |
128.8 |
1502.93(25.1mins) |
可以看到对于长度在1000以内的蛋白序列,结构预测的完整时间基本在半小时以内。这意味着我们用这台8卡A100的服务器,一天时间可以完成至少384个蛋白序列的预测任务。这对于绝大多数的研究团队的计算需求来说都是足够的。当然如果需要更大批量的实现蛋白结构序列的预测,AlphaFold2的整个流程中还有很多的优化空间,比如拆分数据准备阶段和模型预测阶段,把数据准备阶段的多个比对任务并行化等等。这些方法都能进一步提升计算通量。
AlphaFold2模型预测部分的计算过程截图如下:
模型最终的预测结果如下图所示:
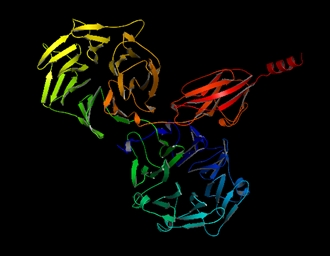
alphaFold2预测的T1050蛋白结构渲染图
2. NAMD测试
NAMD是UIUC开发的一款被广泛使用的分子动力学模拟软件,有十多年的历史。但它不是一个古董软件,这些年一直在进步和优化,特别是在使用GPU做计算性能优化上,NAMD是最早使用GPU 进行计算加速的生命科学计算应用之一。NAMD 采用“time stepping”算法来及时传播分子系统的运动。每个时间步长通常为1或2飞秒,一般来说,实际需要观测的现象通常发生在纳秒或者微秒的尺度上,所以一次有效的模拟往往需要执行数百万个时间步,也就是数百万次的迭代计算。
NAMD的一个时间步通常包含四个主要计算组成部分。如下图所示:
1. 短程非键合力:设置截断半径,对原子力和能量的评估在指定的截止范围 内,属于计算密集型操作,占据 ≈ 90% 的整体 FLOPS。
2. 通过粒子网格 Ewald (PME) 方法计算长程力:在倒易空间利用快速傅里 叶变换计算长程静电相互作用,其中涉及的傅立叶变换 ≈5% 整体 FLOPS。
3. 键合力(成键相互作用):来自分子拓扑结构,占整体 4% 的 FLOPS。
4. 数值积分:计算得到的力, 通过”velocity–verlet” 算法来更新原子速度, ≈1% 整体 FLOPS。
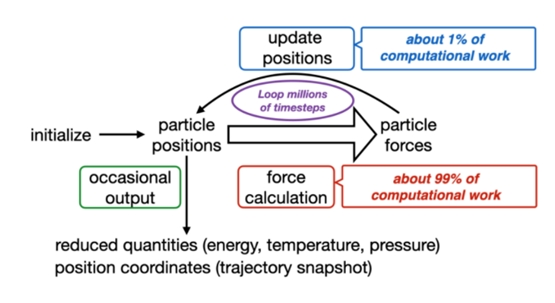
NAMD单个时间步长分解
在 NAMD v2.x 版本中,GPU加速的做法是将所有力的移植到 GPU计算,更新完毕后将数据传回 CPU 主机,并在CPU 端进行数值积分。在这种策略下,NAMD在 CPU和GPU端都存在计算过程,并且需要CPU和GPU之间持续进行数据通信。这种并行策略适用于早期CPU和GPU算力差距还没有特别大的时候,在当前Ampere等新的计算架构,GPU的算力是CPU的百倍以上时,为了大限度的利用GPU算力,NAMD的优化策略从传统的“GPU 加速”优化策略转向完全的“GPU 驻留”策略的转变,即近乎所有的计算都在GPU端完成。
目前 NAMD V3.0 版本不支持源代码编译,因此测试中使用官方网站提供的二进制程序Linux-x86_64-netlrts-smp-CUDA-SingleNode测试,如下图所示。

在运行前,需要在算例的设置文件 *.namd中对应参数进行设置,运行命令为:
|
$ namd3 +p8 +setcpuaffinity +pemap 0 7 +idlepoll +isomalloc_sync ./stmv_nve_cuda.namd +devices 0,1,2,3,4,5,6,7 |
本次测试使用了NAMD自带的卫星烟草花叶病毒 (STMV)算例来进行评测。这个算例是 NAMD 做 benchmark 最常用的算例,含有106万原子,是一种小型二十面体植物病毒,如下图所示。它会使烟草花叶病毒 (TMV) 的感染症状恶化。SMTV是研究分子动力学的绝佳候选者,因为在病毒中它相对较小,在工作站或小型服务器上可以使用传统分子动力学模拟的对象中处于中高级别。测试使用的计算步长为2fs。
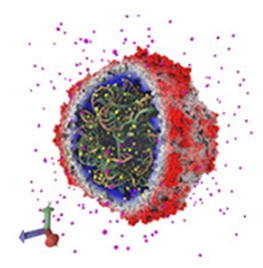
NAMD 测试算例STMV 卫星烟草花叶病毒(包含106,6628 原子)
我们测试了NF5468A5配置1~8块A100 40GB GPU的性能,单位是ns/day,结果如下图所示。实测表明STMV算例在单个A100上可以实现15.6ns/day的计算速度,使用这台服务器全部8个A100可以实现90.6ns/day的计算速度,也就是约11天的时间就能实现1us时间长度的卫星烟草花叶病毒动力学模拟。NAMD使用多卡计算时,并行加速比非常高,8卡并行计算时性能约为单卡的5.81倍,并行效率达到了72.7%左右。对于绝大部分规模在100万原子以内、模拟时长在1us以内的模拟任务,使用单台服务器基本上一周时间就能完成。这对于加速我们的科研工作来说,是非常可观的。
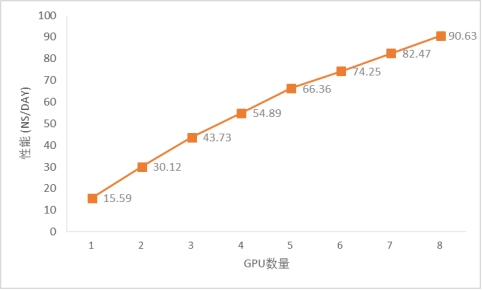
NAMD在NF5468A5平台的测试结果
我们找到了相同算例在CPU平台的测试结果。该平台配置两颗CPU共128核心(单颗64核心、频率2.45GHz),我们使用的NAMD可执行程序为NAMD_2.15alpha1_Linux_x86_64_multicore,最终结果为2.70 ns/day。相比之下, NAMD 在单个A100 GPU上的性能是该CPU平台的5.77倍!假设NAMD在CPU平台集群能够按照线性加速比并行,那么需要使用大约740颗CPU核心的计算集群才能达到一块A100的性能。
三、测试总结
总的来说,浪潮NF5468A5的使用体验还是相当棒的。对于AlphaFold2蛋白结构预测来说,绝大部分蛋白的预测时间都能控制在30min以内,这个速度是相当可观的。对于分子动力学模拟来说,一台服务器1天时间就能实现100万原子近100ns的模拟,这意味着使用一台或者几台NF5468A5 GPU服务器,就能实现计算任务的夕发朝至,晚上下班前提交作业任务,早上上班就能拿到计算结果,工作效率得到大幅提升。
当前我们已经看到AI方法在很多科学计算领域取得了应用和突破,除了AlphaFold2,基于AI方法加速的分子动力学应用DeepMD实现在1天时间内完成1亿原子的第一性原理(ab initio)动力学模拟,其计算规模相比于传统算法提升了100倍。 因此,AI算法的引入,为我们使用分子动力学方法研究更大的体系提供了可能。而AI方法对于科学计算领域的应用加速,不仅仅来自于算法的创新,也来自于AI领域芯片架构和计算加速的创新,像GPU就为AI计算专门设计了TensorCore计算单元。随着AI加速芯片领域的繁荣发展,这些新的技术也会为科学研究提供非常大的帮助。可以预见,以GPU加速为主的异构计算将会成为AI+Science领域的主流硬件平台。